Seht euch nur dieses kleine, unschuldige Protein an. Wenn sich jemand überhaupt für β-2-Mikroglobulin (β2M) interessiert, dann nur, weil es sich in den Gelenken von Dialysepatienten ablagert, denn ohne Nieren bekommt man es nicht ohne weiteres aus dem Blutstrom gefiltert.
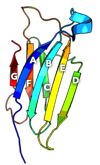
Das strukturelle Motiv von β2M ist wirklich nichts exotisches, ganz im Gegenteil: der immunoglobulin fold findet sich bei unzähligen anderen Proteinen des Immunsystems wieder. Antikörper etwa bestehen aus zwölf dieser Einheiten. Die eigentliche Aufgabe von β2M im Körper besteht darin, zusammen mit einem weiteren Protein einen MHC-Klasse-I Komplex zu bilden. Ohne β2M fällt dieser auseinander und kann seine Funktion nicht mehr erfüllen -- es ist also mindestens ein sehr wichtiges strukturgebendes Element.
Aber war es das schon?
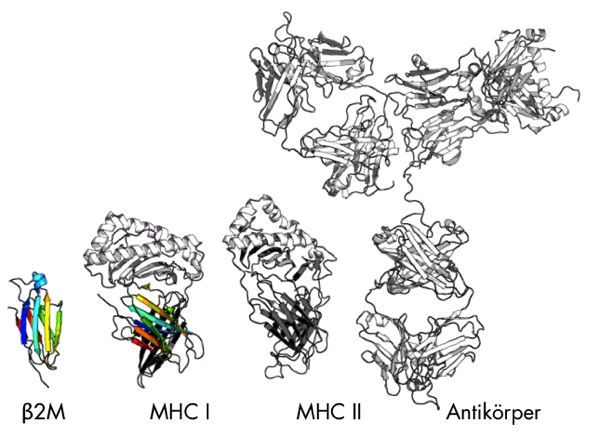
Gerade einmal 100 Aminosäuren lang, besteht β2M aus zwei antiparallelen β-Faltblättern. Die Struktur ist weit verbreitet und entspricht einem grundlegenden Baustein vieler Immunproteine: Der Immunglobulin-Domäne. Sie ist homolog zum zweiten „Beinchen“ des MHC-I-Komplexes (hier in dunkelgrau), entspricht den beiden grau markierten Domänen des nah verwandten MHC-II-Komplexes, und im Antikörper findet sie sich ganze zwölf Mal.
MHC-Komplexe sind dynamische Signalgeber für das Immunsystem
Das Immunsystem kann eigentlich nicht in die Zellen des Körpers hineinsehen, und hier kommen MHC-Komplexe ins Spiel: In einer dafür ausgeformten Tasche binden sie Proteinfragmente von Krankheitserregern oder krankhaft veränderten Proteinen, wie sie etwa bei Krebszellen vorkommen. Die fertigen Komplexe wandern an die Außenseite der Zelle und machen die Proteinfragmente so für das Immunsystem erst zugänglich. T-Zellen des Immunsystems können dann mit ihrem T-Zell-Rezeptor binden, erkennen körperfremde Fragmente und töten virusinfizierte und krankhaft veränderte Zellen ab. Wie diese „Immunsynapse" genau funktioniert, ist noch nicht vollständig verstanden. Sie funktioniert auch leider nicht perfekt, weshalb Menschen und Tiere an Krebs erkranken oder an Autoimmunerkrankungen leiden, die sich auf MHC-Gene zurückführen lassen.
Nicht nur die Form, sondern auch die interne Beweglichkeit und Verformbarkeit der Oberfläche der MHC-Proteine sind für eine erfolgreiche Bindung an die T-Zell-Rezeptoren entscheidend. Wenn beide Oberflächen sehr beweglich sind, können sie sich vielleicht sehr gut aneinander anpassen, allerdings wird die Bindung nicht sehr langlebig sein. Wären die Oberflächen zu starr, könnte der T-Zell-Rezeptor nur wenige Strukturen erkennen und binden -- dabei ist aber eine breite Erkennungsspezifität wünschenswert, um möglichst viele unterschiedliche Erreger aufspüren zu können.
Es ist schwierig, diese dynamischen Vorgänge und das damit zusammenhängende Bindungsverhalten zu erforschen, weil es so wenige Methoden gibt, mit denen man Dynamiken mit entsprechender Auflösung untersuchen kann. Dabei ist es wichtig, dass wir mehr darüber erfahren: peptidbasierte Impfstoffe etwa gegen Hautkrebs befinden sich derzeit in klinischen Tests, aber um ihre Funktion (oder auch Nicht-Funktion) zu verstehen, müssen wir die Immunsynapse besser verstehen. Auch beim Verständnis der molekularen Ursachen bestimmter Autoimmunkrankheiten kann Information über interne Proteindynamik weiterhelfen.
Dynamik konkret
Lässt man T-Zell-Rezeptoren und dazu passende MHC-Komplexe im Reagenzglas aneinander binden, werden die Kontaktflächen unbeweglicher, das ist nicht weiter überraschend. Aber auch relativ weit entfernte Regionen des Proteins verändern ihre Dynamik -- als wenn sich das ganze Protein nach der Bindung versteift. Das betrifft auch Regionen, die vom CD8-Korezeptor der T-Zelle erkannt werden. Dieser Korezeptor hilft der T-Zelle bei der Bindung an den MHC-Komplex. Möglicherweise beeinflusst das dynamische Geschehen zwischen T-Zell-Rezeptor und MHC auch die Bindeeffizienz am anderen Ende des Proteins zwischen CD8 und MHC-Komplex -- eine Signalweiterleitung durch eine veränderte molekulare Dynamik also.
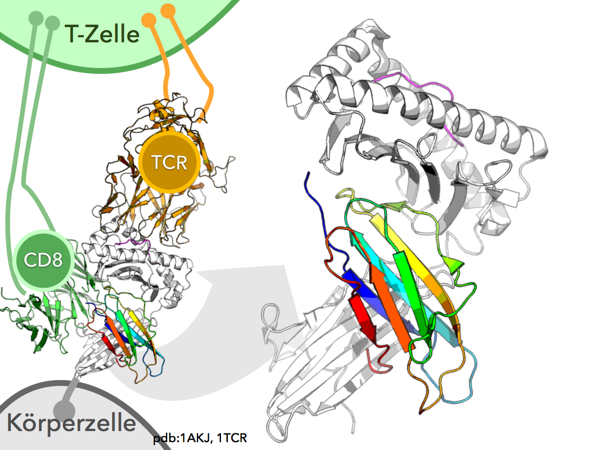
*Links: MHC-Komplexe werden von Immunzellen mit ihren Rezeptoren (hier T-Zell-Rezeptor (TCR) und CD8-Corezeptor) gebunden. Rechts: Der MHC-Komplex in Großaufnahme. Das Proteinfragment, das vom TCR erkannt werden soll, ist in magenta dargestellt.
β2M verändert sich abhängig von MHC-Typ und gebundenem Peptid
Meine Arbeitsgruppe schaut sich MHC-Komplexe und β2M nun schon seit einiger Zeit genauer mit Kernmagnetresonanz (NMR)-Spektroskopie an -- einer Methode, mit der man molekulare Beweglichkeiten indirekt mit atomarer Auflösung messen kann. Am einfachsten geht das, indem man sich die Signale im NMR-Spektrum anschaut, das Unterschiede von subatomaren Magnetfeldern innerhalb des Proteins sichtbar macht. Jedes Atom im Protein hat ein charakteristisches Magnetfeld um sich herum, und über die Position des Signals im Spektrum kann man einzelne Aminosäuren identifizieren. Die Intensität des Signals gibt erste Hinweise auf die Beweglichkeit. Wechselt das Atom etwa langsam zwischen zwei Positionen, sieht man zwei Signale, nämlich für jede Position eine. Werden die Positionen sehr schnell getauscht, dann fallen die beiden Signale zusammen und man sieht nur noch ein einziges Signal. Liegt die Beweglichkeit irgendwo dazwischen, werden die Signale schwächer und sie können sogar verschwinden. Wir haben festgestellt, das bei β2M allein in einer wässrigen Lösung einige Signale völlig verschwinden -- das ist ein starkes Indiz für erhöhte Beweglichkeit.
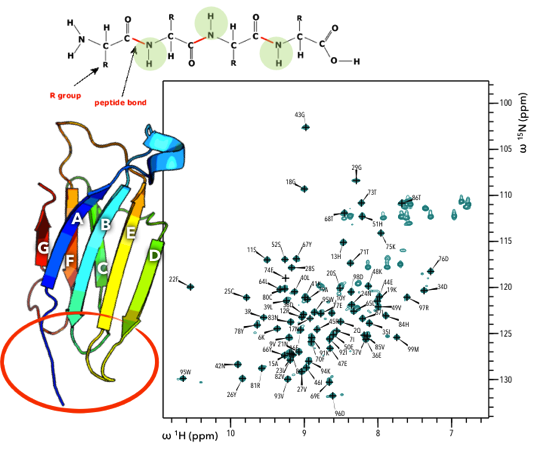
Oben: das Rückgrat eines kurzen Proteins. Grün markiert sind die Atome, die im 1H/15N-Spektrum rechts ein Signal erzeugen. Links: Am β2M sind die Regionen markiert, die sich im Spektrum rechts nicht wiederfinden – genau die Stelle, an denen β2M den Kontakt zum „Hauptprotein” herstellt.
Sobald man sich β2M aber gebunden am MHC-Komplex anschaut, kann man fast alle Signale sehen. Wenn das Protein an seinen natürlichen Bindungsstelle im MHC-Komplex bindet, verliert es also seine ausgeprägte Beweglichkeit.
Dass es trotzdem eine Plastizität an dieser Stelle gibt, konnten wir durch den Vergleich von verschiedenen Komplexen zeigen. In Abhängigkeit von MHC-Protein und gebundenem Peptid verändert sich das Signal von Tryptophan an der Position 60 so stark seine Position, dass wir so sogar vorhersagen könnten, um welchen Komplextyp es sich handelt. Das lässt sich nur teilweise durch die unterschiedliche Aminosäuresequenz von MHC-Bindepartner und gebundenem Proteinfragment erklären, es muss subtile Unterschiede in der Struktur geben, die an das β2M durchgereicht werden.
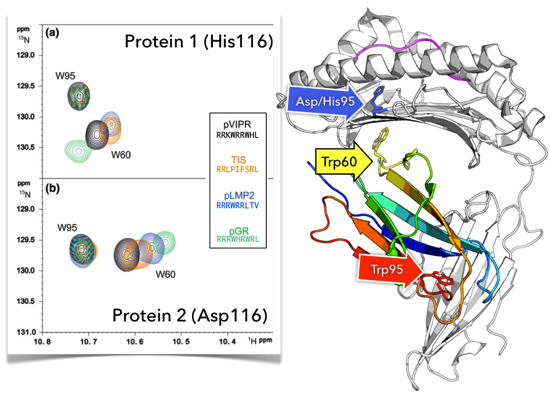
Wie verändern sich β2M-Signale, wenn sich in der Bindetasche etwas ändert? Links: Positionen der Signale der beiden Tryptophan-Seitenkettenamide. Tryptophan 95 (W95) bleibt recht unbeeindruckt, schließlich ist es auch sehr weit entfernt und eher in das Innere von β2M orientiert (siehe recht, roter Pfeil). Tryptophan 60 dagegen liegt direkt in der flexiblen Region und reagiert sehr sensibel auf Veränderungen des Hauptproteins bzw., was in der Bindetasche herumliegt. Abbildung links modifiziert nach [1].
Die spannende Frage ist natürlich, ob die gebundenen Proteinfragmente auch die Dynamik des β2M verändern. Bisher haben wir darauf allerdings noch keine Antwort (aber wir arbeiten dran!). Die Frage, ob β2M also wirklich zu Unrecht als Gerüstprotein vernachlässigt wird, bleibt vorerst noch offen: Unsere Daten zeigen aber, dass β2M immerhin so flexibel zu sein scheint, dass es strukturelle Unterschiede wahrnimmt, die recht weit entfernt stattfinden.
Literatur
1. Beerbaum M, Ballaschk M, Erdmann N, Schnick C, Diehl A, et al. (2013) NMR spectroscopy reveals unexpected structural variation at the protein--protein interface in MHC class I molecules. J Biomol NMR 57: 167--178. doi:10.1007/s10858-013-9777-z.
2. Hee CS, Beerbaum M, Loll B, Ballaschk M, Schmieder P, et al. (2013) Dynamics of free versus complexed β(2)-microglobulin and the evolution of interfaces in MHC class I molecules. Immunogenetics 65: 157--172. doi:10.1007/s00251-012-0667-4.
3. Baker BM, Scott DR, Blevins SJ, Hawse WF (2012) Structural and dynamic control of T-cell receptor specificity, cross-reactivity, and binding mechanism. Immunol Rev 250: 10--31. doi:10.1111/j.1600-065X.2012.01165.x.
